
Yamu
今回は2つのデータセットに
ロジスティック回帰モデルに
当てはめてグラフを観察します
[PR]※本サイトにはプロモーションが含まれています
今回使用するデータセット
1.殺虫剤の濃度と死亡率
| 殺虫剤の濃度 x | 0.4 | 0.57 | 0.7 | 0.8 | 1 |
| 虫の数 | 51 | 48 | 46 | 49 | 50 |
| 死亡数 | 6 | 16 | 24 | 42 | 44 |
| 死亡率 | 0.12 | 0.33 | 0.522 | 0.857 | 0.88 |
下記コードでpythonにデータセットしておきます
import pandas as pd
# データの定義
data1 = {
'殺虫剤の強さ': [0.4, 0.57, 0.7, 0.8, 1],
'虫の数': [51, 48, 46, 49, 50],
'死亡数': [6, 16, 24, 42, 44],
'死亡率': [0.12, 0.33, 0.522, 0.857, 0.88]
}
# データフレームの作成
df1 = pd.DataFrame(data1)
# データフレームの表示
print(df1) 殺虫剤の強さ 虫の数 死亡数 死亡率
0 0.40 51 6 0.120
1 0.57 48 16 0.330
2 0.70 46 24 0.522
3 0.80 49 42 0.857
4 1.00 50 44 0.8802.肺疾患と喫煙データ
| 喫煙本数 x | 2 | 6 | 6 | 10 | 14 | 19 | 22 | 27 |
| 肺疾患の有無 y | 0 | 0 | 0 | 0 | 1 | 0 | 1 | 1 |
下記コードでpythonにデータセットしておきます
import pandas as pd
# データの定義
data2 = {
'喫煙本数': [2, 6, 6, 10, 14, 19, 22, 27],
'肺疾患の有無': [0, 0, 0, 0, 1, 0, 1, 1]
}
# データフレームの作成
df2 = pd.DataFrame(data2)
# データフレームの表示
print(df2)
喫煙本数 肺疾患の有無
0 2 0
1 6 0
2 6 0
3 10 0
4 14 1
5 19 0
6 22 1
7 27 1ロジスティック回帰モデル
2つのデータセットに
ロジスティック回帰モデルを適応してみる
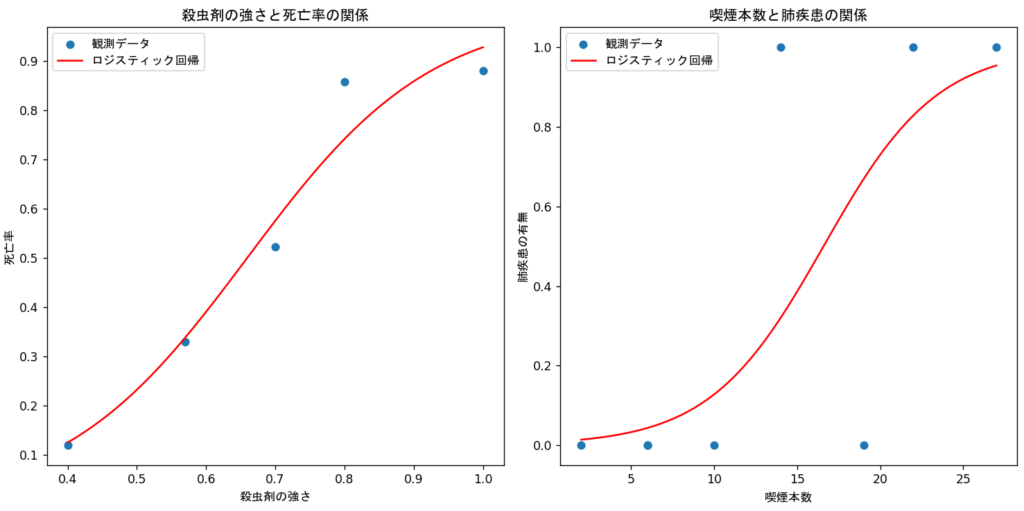
殺虫剤と死亡率のデータに対し
ロジスティック回帰モデルは
当てはまっているように見える
肺疾患の有無のデータでは
煙草の本数が増えるほど
肺疾患を持っている人が確認できているので
1日に何本吸ったら肺疾患になる確率は
何%といった診断ができるかもしれません
下記のコードでデータから
ロジスティック回帰モデルを実装しました
import pandas as pd
import statsmodels.api as sm
import numpy as np
import matplotlib.pyplot as plt
# データの定義
data1 = {
'殺虫剤の強さ': [0.4, 0.57, 0.7, 0.8, 1],
'虫の数': [51, 48, 46, 49, 50],
'死亡数': [6, 16, 24, 42, 44],
'死亡率': [0.12, 0.33, 0.522, 0.857, 0.88]
}
data2 = {
'喫煙本数': [2, 6, 6, 10, 14, 19, 22, 27],
'肺疾患の有無': [0, 0, 0, 0, 1, 0, 1, 1]
}
# データフレームの作成
df1 = pd.DataFrame(data1)
df2 = pd.DataFrame(data2)
# グラフの設定
fig, axs = plt.subplots(1, 2, figsize=(12, 6))
# 日本語フォントの設定
plt.rcParams['font.family'] = 'MS Gothic'
# 1つ目のグラフ: 殺虫剤の強さと死亡率
# ロジスティック回帰モデルの作成
X1 = sm.add_constant(df1['殺虫剤の強さ']) # 切片項を追加
y1 = df1['死亡率']
logit_model1 = sm.Logit(y1, X1)
result1 = logit_model1.fit()
# 殺虫剤の強さの範囲で予測を行う
X_pred1 = np.linspace(df1['殺虫剤の強さ'].min(), df1['殺虫剤の強さ'].max(), 100)
X_pred1 = sm.add_constant(X_pred1)
y_pred1 = result1.predict(X_pred1)
# 散布図と予測結果のプロット
axs[0].scatter(df1['殺虫剤の強さ'], df1['死亡率'], label='観測データ')
axs[0].plot(X_pred1[:, 1], y_pred1, color='red', label='ロジスティック回帰')
axs[0].set_xlabel('殺虫剤の強さ', fontname="MS Gothic")
axs[0].set_ylabel('死亡率', fontname="MS Gothic")
axs[0].set_title('殺虫剤の強さと死亡率の関係', fontname="MS Gothic")
axs[0].legend()
# 2つ目のグラフ: 喫煙本数と肺疾患の有無
# ロジスティック回帰モデルの作成
X2 = sm.add_constant(df2['喫煙本数']) # 切片項を追加
y2 = df2['肺疾患の有無']
logit_model2 = sm.Logit(y2, X2)
result2 = logit_model2.fit()
# 喫煙本数の範囲で予測を行う
X_pred2 = np.linspace(df2['喫煙本数'].min(), df2['喫煙本数'].max(), 100)
X_pred2 = sm.add_constant(X_pred2)
y_pred2 = result2.predict(X_pred2)
# 散布図と予測結果のプロット
axs[1].scatter(df2['喫煙本数'], df2['肺疾患の有無'], label='観測データ')
axs[1].plot(X_pred2[:, 1], y_pred2, color='red', label='ロジスティック回帰')
axs[1].set_xlabel('喫煙本数', fontname="MS Gothic")
axs[1].set_ylabel('肺疾患の有無', fontname="MS Gothic")
axs[1].set_title('喫煙本数と肺疾患の関係', fontname="MS Gothic")
axs[1].legend()
# グラフの表示
plt.tight_layout()
plt.show()モデルの評価
推定されたモデルは実データと
当てはまりの良さを評価する必要があります
評価を行うための指標を紹介します
残差逸脱度
推定したモデルが完全に当てはまったモデルから
どれだけ離れているか数値化したものである
残差逸脱度が大きい場合
モデルがデータを十分に説明していない可能性があるため
モデルの改善が必要です
残差逸脱度は以下のように計算されます
\(D(y,\hat y) = -2log(\frac{L(\hat y)}{L(y)})\)

Yamu
最尤逸脱度は
モデルが
データに対して
適切なものか
評価するための指標なんですね


