
Yamu
今回はRを使って
ロジスティック回帰を実装
その後指標を確認して
モデルを評価します
[PR]※本サイトにはプロモーションが含まれています
今回使うデータセット
1.殺虫剤の濃度と死亡率
| 殺虫剤の濃度 x | 0.4 | 0.57 | 0.7 | 0.8 | 1 |
| 虫の数 | 51 | 48 | 46 | 49 | 50 |
| 死亡数 | 6 | 16 | 24 | 42 | 44 |
| 死亡率 | 0.12 | 0.33 | 0.522 | 0.857 | 0.88 |
下記コードをrに書き込みデータセットします
# 濃度
x = c(0.4, 0.57, 0.7, 0.8, 1)
# 虫の数
m = c(51, 48, 46, 49, 50)
# 死亡数
y = c(6, 16, 24, 42, 44)
# 死亡率
p = y/m
# データフレームの作成
data = data.frame(x, m, y, p)
# データフレームの表示
print(data)
実行結果
x m y p
1 0.40 51 6 0.1176471
2 0.57 48 16 0.3333333
3 0.70 46 24 0.5217391
4 0.80 49 42 0.8571429
5 1.00 50 44 0.8800000Rでロジスティック回帰モデルを実装する
殺虫剤の強さ(濃度)と
死亡率を散布図に
プロットするコードを書きます
plot(x, p, xlim =c(0,1.2), ylim =c(0, 1), pch =19, cex =1.5, cex.lab =1.5)実行結果
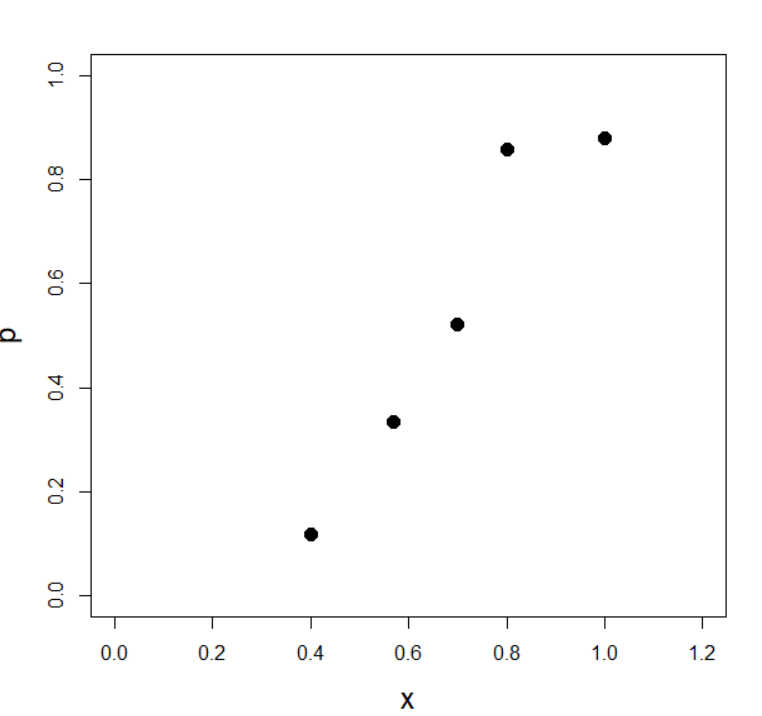
plotのオプションの説明をします
| オプション | コードの意味 |
| plot(x,y) | 2つのベクトルをプロット |
| xlim | x軸の範囲 |
| ylim | y軸の範囲 |
| pch | プロットの形状を示す |
| cex | プロットするサイズ |
| cex.lab | x軸とy軸のラベルのサイズを指定 |
ロジスティック回帰分析を行う
コードを紹介します
res = glm(cbind(y, m - y) ~ x, family = "binomial", data = data)
rescbind(y, m -y)によって
二項分布における成功と失敗のペアを持つ行列が生成されます
ここでいう成功と失敗は
虫の死亡数と生存数です
family = “binomial”で回帰モデルを生成します
実行結果
Call: glm(formula = cbind(y, m - y) ~ x, family = "binomial", data = data)
Coefficients:
(Intercept) x
-4.940 7.497
Degrees of Freedom: 4 Total (i.e. Null); 3 Residual
Null Deviance: 98
Residual Deviance: 5.881 AIC: 29.1Coeffocoentsに回帰係数
Null Devianceに最大逸脱度
Residual Devianceに残差逸脱度
一般的に、モデルの適合度が良い場合
Residual Deviance は Null Deviance
よりもかなり小さい値になります
Null Deviance と Residual Deviance の差が大きいほど
予測変数が応答変数をうまく説明していることを示しています。
このモデルはResidual Devianceがとても小さい値になっているので
予測変数 x が応答変数 y を
非常によく説明していることを示しています
ロジスティック回帰をプロットします
x_seq <- seq(min(data$x), max(data$x), length.out = 100)
y_pred <- predict(res, newdata = data.frame(x = x_seq), type = "response")
lines(x_seq, y_pred, col = "red", lwd = 2)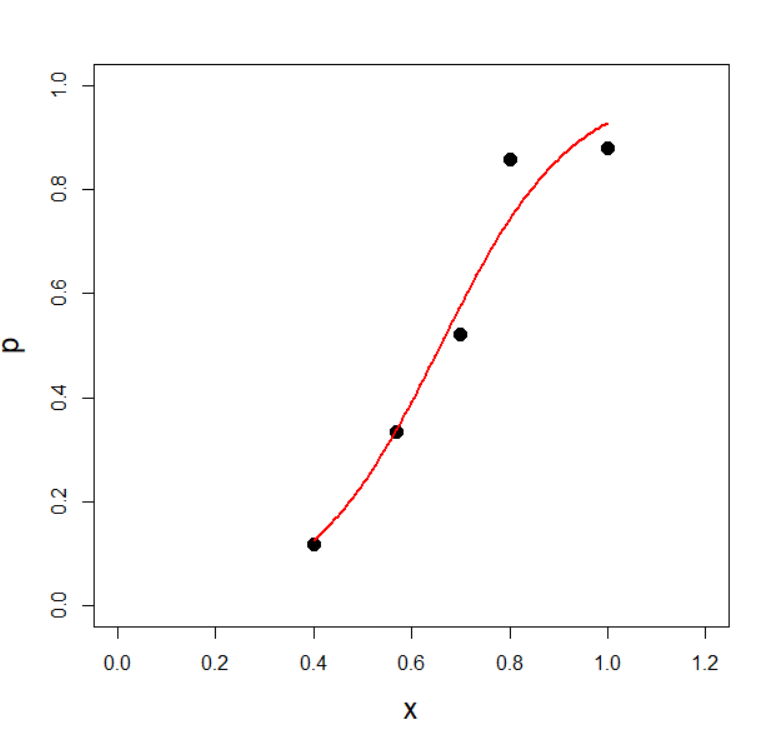
最後にコードを纏めておきます
# 濃度
x = c(0.4, 0.57, 0.7, 0.8, 1)
# 虫の数
m = c(51, 48, 46, 49, 50)
# 死亡数
y = c(6, 16, 24, 42, 44)
# 死亡率
p = y/m
# データフレームの作成
data = data.frame(x, m, y, p)
# データフレームの表示
print(data)
plot(x, p, xlim =c(0,1.2), ylim =c(0, 1), pch =19, cex =1.5, cex.lab =1.5)
res = glm(cbind(y, m - y) ~ x, family = "binomial", data = data)
res
x_seq <- seq(min(data$x), max(data$x), length.out = 100)
y_pred <- predict(res, newdata = data.frame(x = x_seq), type = "response")
lines(x_seq, y_pred, col = "red", lwd = 2)

